研究紹介 <研究紹介ビデオはこちら>
現在進行中の主な研究内容
siRNA&CRISPR研究
(1) 疾患関連遺伝子に対する特異的RNA干渉法の開発とその臨床応用
(1) 疾患関連遺伝子に対する特異的RNA干渉法の開発とその臨床応用
(関連論文:Kobayashi et al. 2021, Iribe et al. 2017; Ui-Tei et al. 2008)
(2) RNA干渉における塩基対合の熱力学的制御と化学修飾による効果の解明
(2) RNA干渉における塩基対合の熱力学的制御と化学修飾による効果の解明
(関連論文:Tian et al. 2019; Ui-Tei. 2016; Hibio et al. 2012; Ui-Tei et al. 2012; Ui-Tei et al. 2008)
(3) CRISPR/Cas9システムによるゲノム編集の分子メカニズム解明とゲノムワイドな応用技術開発
(関連論文:Ui-Tei et al. 2017; Naito et al. 2015)
(3) CRISPR/Cas9システムによるゲノム編集の分子メカニズム解明とゲノムワイドな応用技術開発
(関連論文:Ui-Tei et al. 2017; Naito et al. 2015)
miRNA研究
(4) マイクロRNAが制御するヒトの新たな生体防御機構とその遺伝子ネットワーク全貌の解明
(5) A-to-I RNA editing酵素などのRNA結合タンパク質によるエピトランスクリプトーム研究
(6) miRNAサイレンシング因子TNRC6Aの細胞核内機能の解明と細胞がん化との関連
これまでの主な研究内容
ノンコーディングRNAの機能解析はゲノム科学の大きな課題
ゲノム配列解読プロジェクトにより、いわゆるタンパク質をコードする“遺伝子”の数は、下等生物から高等生物に至るまで、大きくは異ならないことがわかってきました。生命現象はゲノム情報によって制御されていると考えられているにも関わらず、下等生物とヒトの遺伝子数が大きく変わらないとすれば、思考や感情あるいは言語などの複雑な高次生命機能は、いかにして生み出されるのでしょうか。近年のトランスクリプトーム解析によって、ヒトを含む高等真核生物にはタンパク質をコードしないノンコーディングRNA(non-coding RNA)と呼ばれるRNAが多く存在することが明らかになってきましたが、ノンコーディングRNAの数や多様さと生物の複雑さには強い相関が見られることから、これらが生物の高次機能に深く関与している可能性が指摘されており、ノンコーディングRNAの機能を明らかにすることは、ゲノム科学における大きな課題となっています。
さまざまな環境要因がRNAを介して遺伝情報を制御する
私たちの体は細胞という生命の最も基本的な構成単位からなりたっています。細胞内ではゲノムを構成するDNAからRNAが転写され、RNAからタンパク質が翻訳されるといった、セントラルドグマと呼ばれるゲノムの遺伝情報を読み解く流れが存在し、それによって生命体が機能します。しかし、ゲノムプロジェクトがすすみ、ゲノム情報のもつ全体像が明らかになるにつれて、生命機能はゲノムだけで決まってくるのではなく、環境に由来する要因も重要であることが明らかになってきました。すなわち、複雑で高次な生命現象や加齢に伴う遺伝子発現の変動は、光、重力、放射線や温度、さらには微生物の感染やストレスといった環境要因が大きな影響をもたらすのです。近年、RNAに関する研究が急速に進展し、RNAは単なる遺伝情報の仲介役ではなく、その柔軟な性質を利用して配列や構造に依存した多様な機能を示すこと明らかになってきました。このようなノンコーディングRNAは、これまで研究対象となっていたRNA群とは異なるものと位置づけられています。また、RNAは様々な化学修飾によってエピトランスクリプトームという新しい遺伝子発現調節の過程を構築する分子としても注目されています。本研究室では、環境の影響をうまく伝える新しい遺伝子制御の機構についてRNAを中心に研究しています。
RNAの作用機序を分子生物学的・生化学的・細胞生物学的に解析し、ゲノムワイドな機序について情報科学的に解析していきます。
1)1遺伝子特異的に効率よくノックダウンできるsiRNA配列設計ガイドライン
RNAi は、ある遺伝子の特定領域と相同な2本鎖RNA (double-stranded RNA, dsRNA) がmRNAの相同部分を切断するという現象です。しかし、多くの哺乳類細胞では、約30塩基対以上の長いdsRNAが細胞内へ入ると、ウイルスに対する生体防御機構であるインターフェロン応答が誘導され、細胞が死に至ります。しかし、私たちは、胚性幹細胞などの限られた細胞系であれば哺乳類でも長いdsRNAによるRNAiが誘導できることを示し、哺乳類細胞にもRNAiのマシナリーは存在することを明らかにしました。
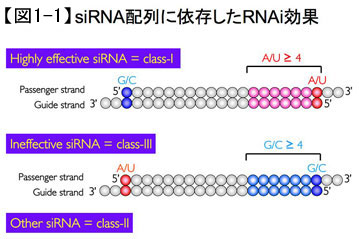
その後、長いdsRNAはDicerというRNaseⅢ酵素によって、3’が2塩基突出した短いsiRNAに切断され、切断産物である短いsiRNAは、哺乳類細胞でもインターフェロン応答を起こさずにRNAiが誘導できることが示されました。RNAi法は操作も簡便で、塩基配列は最低19塩基わかっていればよいため、ポストゲノムシークエンス時代に適した有用な遺伝子機能解析法として注目されています。しかし、哺乳類細胞では、どのような配列のsiRNAでもRNAiを誘導できるわけではなく、RNAiが起こるかどうかはsiRNAの配列に大きく依存していることを私たちは見いだしました。レポーター遺伝子や内在性遺伝子に対する培養細胞を用いたRNAi実験、および個体におけるRNAiやDNAベクター型RNAiなどの種々の実験から、効くsiRNAの配列は、以下の4つの条件をすべて満たすものであると考えられました(図1—1)。
(1)ガイド鎖の5’末端がAまたはUである。 (2)パッセンジャー鎖の5’末端がGまたはCである。 (3)アンチセンス鎖の5’領域にAまたはUが多い。 (4)長いGCの連続配列がない。このような効く配列の条件を満たし、標的以外のすべての遺伝子とは最低2塩基以上のミスマッチのあるsiRNA配列設計用のウエブサイト(siDirect)を構築しました。これは、誰でも利用することが可能なようにWeb上に公開されています。 上記の条件を満たしているsiRNAは、なぜRNAi効果が高いのでしょうか。RNAi効果の高いsiRNAと低いsiRNAでは、図1に示すような顕著な非対称性がありました。RNAiの実行過程において、siRNAは2本鎖がほどけて1本鎖となり、RNA-induced silencing complex (RISC)複合体に取り込まれて標的mRNAと対合します。2本鎖RNAのAまたはUの対合は、GまたはCに比べ不安定であり、AとUの対合の方がほどけやすいという性質があります。したがって、効くsiRNAのガイド鎖の5’領域にAまたはUが多いということは、siRNAは非対称的に、ガイド鎖の5’側から1本鎖へほどけていくであろうことを示唆していました。一方、我々はRISCの中核となるタンパク質であるArgonaute (Ago)のヒト・マウス遺伝子を始めて同定していますが、最近の好熱細菌のAgoの構造解析の研究から、siRNAのガイド鎖の5’末端領域の6塩基はAgoの表面に固定されていることが明らかにされました。Agoに固定されるガイド鎖の末端は、最初に1本鎖になった末端側と考えられることから、前述の条件を満たすsiRNAが効率よくRNAiを誘導できるのは、ガイド鎖が効率よくRISC複合体に取り込まれる配列であるためと考えられました。
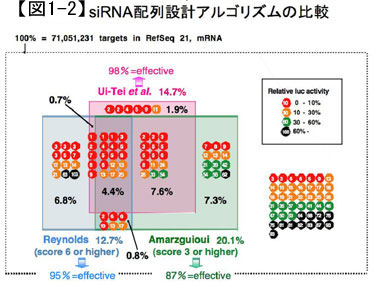
有効なsiRNA配列設計アルゴリズムとしては、私たちのアルゴリズム以外にもいくつか提唱されていますが、私たちの方法を含む、世界的に広く利用されている3つのアルゴリズムについて、その有効性をホタルルシフェラーゼ遺伝子を標的するレポーターアッセイを用いて検討しました。その結果、私たちのアルゴリズムで選択したsiRNAの98%は標的遺伝子のmRNA量を33%以下に抑制するという最も良い結果が得られました(図1—2)。私たちの方法では、ヒト遺伝子の99.5%に対して有効なsiRNA配列が選択できることから、本研究によりヒトを始めとする哺乳類におけるRNAiの基盤的技術が、ほぼ確立できたと考えています。
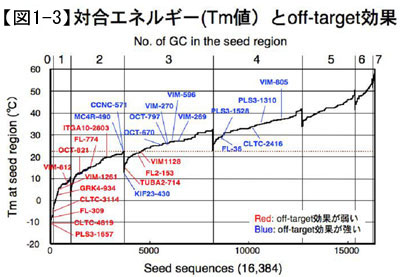
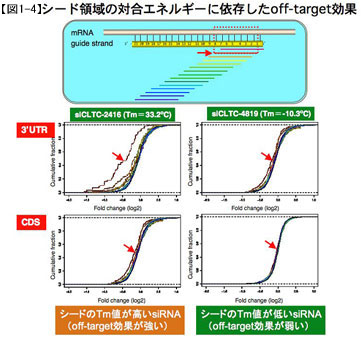
しかしながら、RNAiでは、目的とした遺伝子と完全に相補的な配列のsiRNAを用いているにも関わらず、目的とした遺伝子以外の遺伝子が抑制される場合があることがわかってきました。私たちはどのような機構でoff-target効果が起こるのかをマイクロアレイのデータを用いて解析した結果、siRNAガイド鎖の5'末端から2-8塩基目の7塩基(シード領域と呼ばれる)のRNAのみが相補的な配列をもつ遺伝子はoff-target効果によって抑制されると考えられる結果を得ました。つまり、siRNAの場合、全長にわたって完全に相補的な配列をもつ遺伝子(標的遺伝子)はRNAi効果によって抑制されますが、それだけではなく、シード領域だけが相補的な配列をもつ遺伝子(非標的遺伝子)も、off-target効果と呼ばれる、本来は意図しない作用によって抑制される場合があることになります。RNAi法によって目的とする1遺伝子のみをノックダウンするためには、off-target効果が起こらない方法が必要になりますが、ランダムな7塩基の配列は47=16,384塩基に1回の確率で出現することになりますが、off-target効果はsiRNAの配列によって起こり方が異なるため、配列以外の要因が関わると考えました。すなわち、off-target効果はsiRNAのシード領域と標的mRNAが塩基対合することによって起こるため、この塩基対合の対合エネルギーがoff-target効果の程度を制御する主要な要因であると考え、それを実証する研究を行いました。融解温度(melting temperature, Tm)と自由エネルギー(Gibbs free energy change, ΔG)という2つの対合エネルギーの指標を用いて検討した結果、たった7塩基であっても、2本鎖RNAのTm値は約70℃の大きな違いがあり、予想どおり、シード領域のTm値が高いものはoff-target効果が強く、Tm値が低いものではoff-target効果はほとんど認められませんでした(図1—3)。ΔGでも同様の傾向が認められましたが、Tmを用いたときのほうが、より強い相関が認められました。また、マイクロアレイの結果では、シードと相補的な配列が3’UTRにある遺伝子は強く抑制されますが、CDSにある場合にはその抑制の程度は小さいという違いがありました(図1-4)。
しかし、両者とも、シードのTm値と抑制効果には同様の強い相関が認められたことから、siRNAによるoff-target効果の強さは対合エネルギーに依存しているといえるでしょう。したがって、3’UTRに標的部位があるほうが強い抑制効果が認められる原因は、siRNAそのものにあるのではなくmRNA側にあると考えられました。
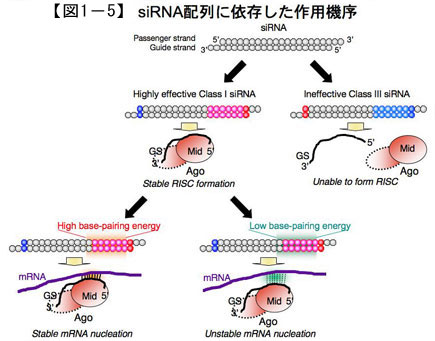
このような研究から、小分子RNAの作用機序におけるsiRNAの配列の寄与の大筋が明らかになってきました(図1-5)。 RNAi効果が高いsiRNA配列の特性は、主としてガイド鎖がRISCへ取り込まれるステップに関わっていると考えられました。さらに、シード領域と標的遺伝子との対合が熱力学的に不安定化することで、off-target効果を避けることが可能であることも明らかとなってきました。このようなTmを考慮したsiRNAの配列設計が可能なウェブサーバーも構築しており、siDirect 2.0として公開しています。
2)siRNAの非対称的な領域特異的役割
私たちは、小分子RNAの作用機序を明らかにするためのアプローチの1つとして、21塩基程度と非常に短いsiRNAの領域ごとの役割の違いを調べました。siRNAの一部をシステマテイックにDNAに置換するという一連の実験を行い、効くsiRNAのガイド鎖の5’末端の約1/3の領域(5’末端から8塩基)はすべてをDNAに置換してもRNAi活性に影響はないが、3’側の残りの約2/3は数塩基でもDNAに置換するとRNAi活性がほとんど消失することを見いだしました。siRNAの5’末端の約1/3の領域は上述した”シード”と呼ばれる領域(5’末端から2−8塩基)とほぼ一致しています。私たちの結果ではシード領域をDNAに置換したsiRNAのRNAi効果はほとんど変化しなかったことから、最終的に全長にわたって対合する遺伝子に対するsiRNAのシード領域の役割は、まず最初に標的の配列を識別して対合することであり、そのためにはシード領域は必ずしもRNAである必要はなくDNAであっても問題ないと考えられました(図2-1)。
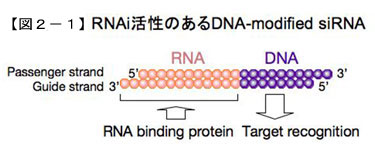
しかし、シードだけが相補的な配列をもつ非標的遺伝子に対しては、シード部分のみが作用点として働くと考えられます。RNA-RNAの対合はRNA-DNAよりも強いため、非標的遺伝子に対する抑制効果は、シードがRNAであるsiRNAの場合には標的遺伝子とRNA-RNA対合をするため強いoff-target効果が起こることが予想されますが、この部分をDNAに置換することによって減弱されると考えられます。一方で、ガイド鎖の3’側は、DNAに置換するとRNAi活性がほぼ完全に消失します。この領域はRLC (RISC-loading complex)あるいはRISCに含まれるRNA結合タンパク質との結合実験から、2本鎖RNA結合タンパク質の結合部位として機能していると考えられました(図2-1)。現在のこの領域のもつ役割について検討中です。
3)ヒト全遺伝子に対するsiRNAライブラリの構築
私たちのsiRNA配列設計ガイドラインに基づき、文部科学省ゲノムネットワークプロジェクト(平成16~20年度)の一環として、東京大学分子細胞生物学研究所の秋山徹教授らと共同で、ほぼすべてのヒト遺伝子をカバーする、約20,000のレトロウイルス型siRNAライブラリを構築しました。さらに、およそ5,000遺伝子に対しては、胚性幹細胞や神経細胞にも適用可能なレンチウイルス型siRNAライブラリも作製しました。しかし、これらのベクターはRNA ポリメラーゼIIIプロモータを使用しているため、プロモータの性質上、選択できるsiRNAの配列が制限され、設計が困難な遺伝子もありました。そこで、独自に開発したRNA ポリメラーゼII プロモータによるベクターを用いて、約2,400遺伝子に対するライブラリを構築しました。約1,300遺伝子についてはヒト/マウス共通配列を用いました。これらのライブラリから転写関連遺伝子約500を選択し、HeLaおよびRKO細胞に導入して内在性遺伝子に対する効果を検討した結果、約75%の遺伝子についてRNAi効果を確認することができました。したがって、このライブラリは世界的にみてもレベルの高い品質を保持している我が国唯一のものであり、実際のヒト全遺伝子機能解析に使用可能な有用なツールといえます。現在、このライブラリは、小分子RNAの作用機序や癌化の研究などに利用されています。
4)miRNAが細胞核へ輸送される分子メカニズム [プレスリリース] [Todai Research]
microRNAは、長さ20数塩基の小さな1本鎖のRNAで、直接相互作用するAgoタンパク質やAgo結合タンパク質であるGW182などとmicroRNA-induced silencing complex (miRISC)と呼ばれる複合体を形成し、相補的な塩基配列をもつmRNAと対合して、その分解や翻訳抑制を引き起こすことが知られています。microRNAによって抑制されるRNAは主に成熟型のmRNAであることや、AgoやGW182が細胞質内の顆粒状構造体であるProcessing body(P body)に局在することなどから、microRNAによる遺伝子の抑制は主として細胞質で起こる現象であると考えられていました。
一方で、核内にもmicroRNAやAgoが存在することや、microRNAやsiRNAといった小分子RNAが、核内で転写レベルで遺伝子発現を制御したり、mRNAのスプライシングを制御することを示唆する報告もなされていました。しかし、microRNAやAgoを含む複合体が、どのように核内に運ばれて機能するのかという分子的な機構については、今日までほとんど分かっていませんでした。
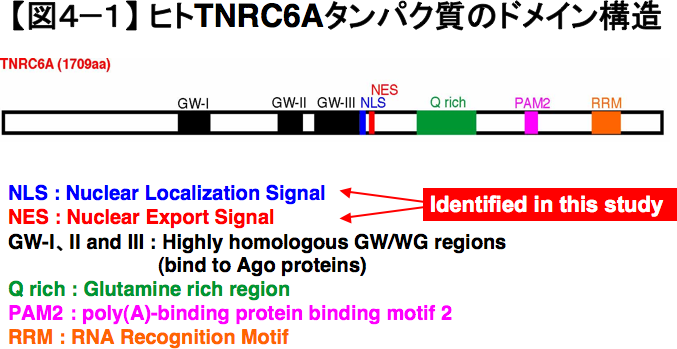
私たちは、microRNAの核内移行の分子機構を明らかにするために、まずmicroRNAと相互作用するタンパク質の細胞内局在を解析しました。特にヒトGW182ファミリータンパク質の一つであるTNRC6Aに着目し、そのさまざまな領域の断片に緑色蛍光タンパク質(Green fluorescent protein, GFP)を融合させたタンパク質をヒト培養細胞に発現させて、局在を解析しました。その結果、TNRC6Aの一次配列には、タンパク質を細胞質から核に移行させるアミノ酸配列である核移行シグナル(Nuclear localization signal, NLS)と、タンパク質を核から細胞質に輸送させるアミノ酸配列である核外移行シグナル(Nuclear export signal, NES)の両方が存在すると考えられる結果が得られました(図4—1)。
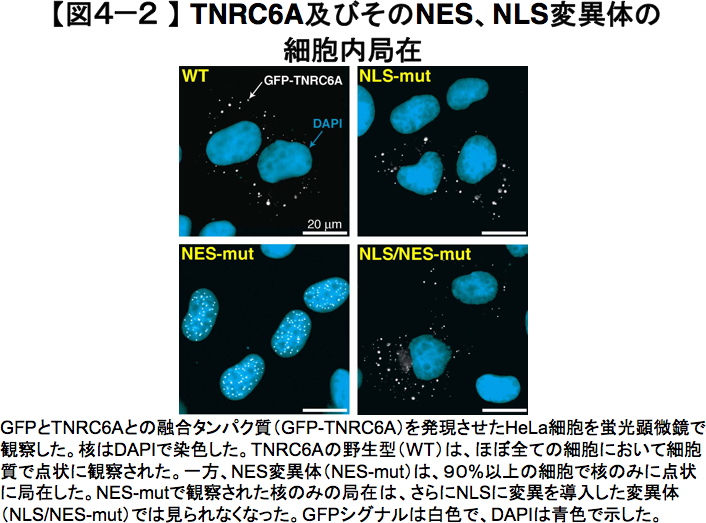
そこで、同定したNESが本当に機能するのかを調べるために、NESのアミノ酸配列に変異を導入し、その細胞内局在を観察しました。すると、ほとんどのTNRC6Aは核内に留まり、きれいな点状の局在を示しました(図4—2)。
また、NESに加えてNLSにも変異を導入すると、このような核移行は見られず、大部分は細胞質に留まることが分かりました。すなわち、TNRC6Aは、私たちが見出したNLSとNESの両方のはたらきによって、核と細胞質の間を行き来する核—細胞質間輸送タンパク質であることが明らかになりました。さらに、TNRC6AのNES変異体が存在する核内の点状構造体には、Agoタンパク質やmicroRNAも共局在していました(図4—3)。
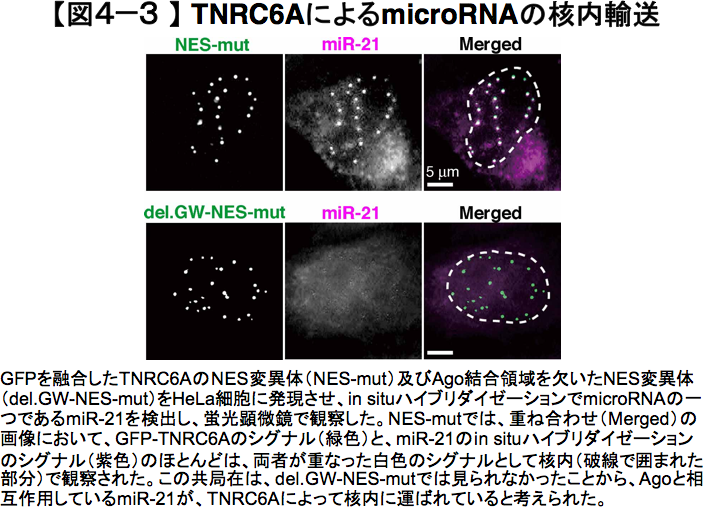
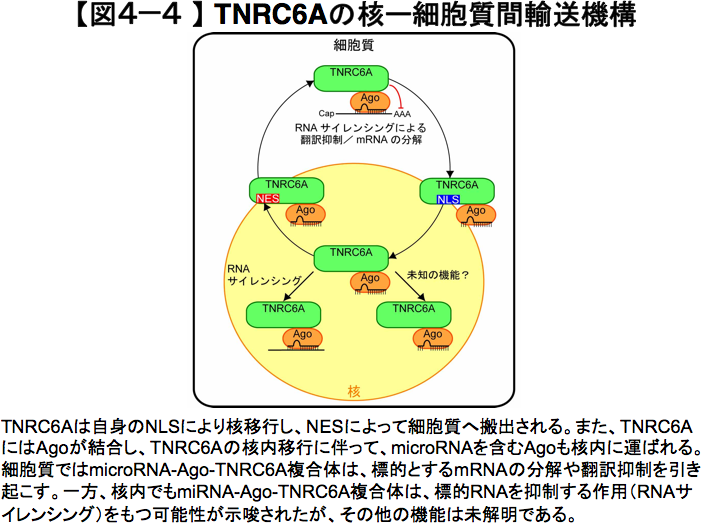
このようなAgoタンパク質やmicroRNAとの共局在は、Ago結合領域を欠いたTNRC6A変異体では見られなかったことから、TNRC6AはAgoとの相互作用を介してmicroRNAを核内に輸送していると考えられました。また、TNRC6Aの核外移行シグナル変異体を強制発現させると、核内非コードRNAとして知られているMALAT-1に対するsiRNAによるノックダウン効果が増強されました。したがって、TNRC6Aは、核内における小分子RNAによるRNAの発現抑制に関わっていると考えられます。 本研究によって、TNRC6Aが小分子RNAとAgoタンパク質を含む複合体を細胞質から核内に輸送することが初めて示され、核内でもmicroRNAによる遺伝子発現調節が行われる可能性が提起されました(図4—4)。
核内microRNA複合体は、核内RNAの発現を抑制する他にも、転写段階での遺伝子発現制御やRNAのスプライシング制御に関わる可能性、さらには全く未知の機能をもつ可能性も考えられます。microRNA-Ago-TNRC6Aを最少単位とする核内複合体の機能を明らかにするために、この複合体に含まれる他のタンパク質分子や標的とするRNAの同定を、現在進めています。
5)miRNAサイレンシングの定量的解析
microRNAは塩基配列特異的な遺伝子サイレンシングの主要な調節因子ですが、microRNAによる標的遺伝子のサイレンシング効率を決定する要因については、よくわかっていませんでした。私たちは、1)でも述べたように、siRNAによるoff-target効果の強さは、シード領域と対合するmRNAとの塩基対合力に依存することを明らかにしていました(図5-1)。
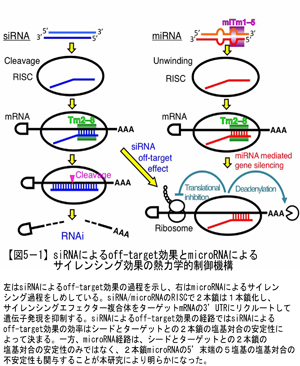 一方で、microRNAによる遺伝子サイレンシングの機構は、siRNAによるoff-targetの機構と非常に良く似ていることから、microRNAによるサイレンシングの効率を数式を用いて定量化することを試みました。その結果、microRNAによるサイレンシングの効率はシード領域とターゲットmRNAとの塩基対合力(Tm2-8)のみによって決まるのではなく、microRNAの2本鎖状態のときの5’末端の5塩基の対合力の安定性(miTm1-5)によっても変動することがわかりました(図5-2)。
一方で、microRNAによる遺伝子サイレンシングの機構は、siRNAによるoff-targetの機構と非常に良く似ていることから、microRNAによるサイレンシングの効率を数式を用いて定量化することを試みました。その結果、microRNAによるサイレンシングの効率はシード領域とターゲットmRNAとの塩基対合力(Tm2-8)のみによって決まるのではなく、microRNAの2本鎖状態のときの5’末端の5塩基の対合力の安定性(miTm1-5)によっても変動することがわかりました(図5-2)。
すなわち、microRNAの5’末端の2本鎖部分が不安定であり、シード領域と標的mRNAによって形成される2本鎖が安定なmiRNAが、強力なサイレンシング活性を示すことを明らかにし、microRNAによる遺伝子サイレンシングの強さは、Tm2-8-0.53×miTm1-5で表すことができることを明らかにしました。この結果は、2本鎖miRNAの2本のRNA鎖のうち、不安定な5’末端を持つRNA鎖は、容易にRNA-induced silencing complex(RISC)に取り込まれること、また、miRNAは、シード領域と相補的な配列を持つmRNAを標的として認識し、抑制するというこれまでの知見と一致します。
しかし、それを数式として定量的に表すことによって、シード領域とターゲットmRNAとの塩基対合力(Tm2-8)に対して、microRNAの2本鎖状態のときの5’末端の5塩基の対合力の安定性(miTm1-5)は約半分の寄与をしていることを明確に示しました。このように、私たちの結果は、miRNAの一本鎖化の過程および標的認識過程の両方が、塩基対合の熱力学的性質によってうまく制御されている可能性を示唆していました。

すなわち、microRNAの5’末端の2本鎖部分が不安定であり、シード領域と標的mRNAによって形成される2本鎖が安定なmiRNAが、強力なサイレンシング活性を示すことを明らかにし、microRNAによる遺伝子サイレンシングの強さは、Tm2-8-0.53×miTm1-5で表すことができることを明らかにしました。この結果は、2本鎖miRNAの2本のRNA鎖のうち、不安定な5’末端を持つRNA鎖は、容易にRNA-induced silencing complex(RISC)に取り込まれること、また、miRNAは、シード領域と相補的な配列を持つmRNAを標的として認識し、抑制するというこれまでの知見と一致します。
しかし、それを数式として定量的に表すことによって、シード領域とターゲットmRNAとの塩基対合力(Tm2-8)に対して、microRNAの2本鎖状態のときの5’末端の5塩基の対合力の安定性(miTm1-5)は約半分の寄与をしていることを明確に示しました。このように、私たちの結果は、miRNAの一本鎖化の過程および標的認識過程の両方が、塩基対合の熱力学的性質によってうまく制御されている可能性を示唆していました。
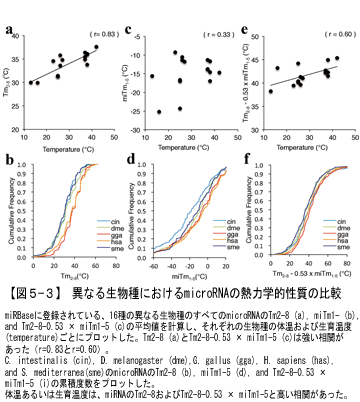
さらに興味深いことに、そのような塩基配列の相違に依存した熱力学的性質の違いがmicroRNAによるサイレンシング効率の決定要因であるとすれば、異なる体温の生物種では、同じmicroRNAが異なる強さのサイレンシング活性を示す可能性があることが推定されます。そこで、体温あるいは生育温度が異なる16種の生物種のすべてのmicroRNAのTm2-8, miTm1-5, およびTm2-8-0.53 × miTm1-5の平均値を算出し、それぞれの生物種の体温および生育温度との相関関係を調べました(図5-3)。すると、Tm2-8およびTm2-8-0.53 × miTm1-5は、それぞれの生物種の体温あるいは生育温度と強い相関関係があることが明らかになりました。したがって、microRNAは進化の過程において、その体温あるいは生育温度によってmicroRNAの選択を受けている可能性が考えられました。